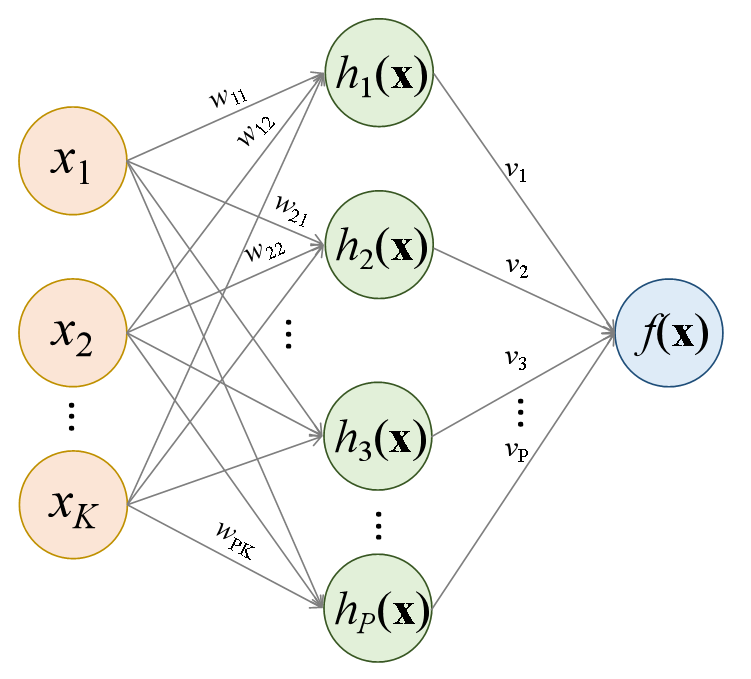
11 Redes neurais para regressão
11.1 Definições iniciais
Seja um vetor de \(K\) variáveis regressoras \(\mathbf{x}=[x_1,x_2,\ldots,x_K]^T\), uma variável dependente ou de resposta contínua, \(y \in \mathbf{R}\) e \(N\) observações de treino disponíveis, \(\{x_1,y_1\}, \{x_2,y_2\}, \ldots, \{x_N,y_N\}\).
Uma rede neural com 1 camada oculta é ilustrada na Figura 11.1. Esta rede apresenta \(K\) variáveis preditoras ou independentes na camada de entrada, \(P\) neurônios na camada oculta, \(h_1(\mathbf{x})\), \(h_2(\mathbf{x})\), …, \(h_P(\mathbf{x})\), e entrega na camada de saída o modelo \(f(\mathbf{x})\).

Começando de trás para frente, o modelo final pode ser definido a partir da Equação 11.1. \[ \begin{matrix} f(\mathbf{x})=v_0 + v_1h_1(\mathbf{x}) + v_2h_2(\mathbf{x}) + \ldots + v_Ph_P(\mathbf{x}) \\ f(\mathbf{x})=v_0 + \sum_{p=1}^Pv_ph_p(\mathbf{x}) = \mathbf{v}^T\mathbf{h} \end{matrix} \tag{11.1}\]
onde \(\mathbf{v} = [v_0, v_1, ..., v_P]^T\) é um vetor de pesos do modelo final na camada oculta e \(\mathbf{h} = [1, h_1(\mathbf{x}), ... h_P(\mathbf{x})]^T\) é um vetor de funções computadas na camada oculta. O \(p\)-ésimo termo \(h_p(\mathbf{x})\), \(p=1, ..., P\), da camada oculta é calculado conforme Equação 11.2. \[ \begin{matrix} h_p=g(w_{p0} + w_{p1}x_1 + w_{p2}x_2 + \ldots + w_{pK}x_K) \\ h_p=g(w_{p0} + \sum_{k=1}^Kw_{pk}x_k) = g(\mathbf{w}_p^T\mathbf{x})\\ \end{matrix} \tag{11.2}\]
onde \(\mathbf{w}_p = [w_{p0}, w_{p1}, \ldots, w_{pK}]^T\) é um vetor de pesos do \(p\)-ésimo neurônio na camada de entrada e \(g(z)\) é uma função de ativação a qual deve ser selecionada de forma apropriada ao tipo de problema abordado. É importante observar que tano na camada oculta quanto na camada de saída não foram ilustrados na Figura 11.1 os termos de vício \(v_0\) e \(w_{0p}\), porém são importantes e considerados no modelo. Para problemas de regressão a função de unidade linear retificada (Retified Linear Unit - ReLU) pode ser usada como ativação nas camadas ocultas, sendo esta descrita matematicamente conforme Equação 11.3.
\[ g(z) = max(0,z) = \bigg\{ \begin{matrix} 0,z<0 \\ z,z\geq0 \end{matrix} \tag{11.3}\]
A função de ativação ReLu é plotada a seguir. Uma vez que no processo de otimização dos parâmetros da rede, o gradiente da função perda em relação a tais parâmetros é computado iterativamente, a função de ativação ReLU apresenta a vantagem de ter derivada nula para valores negativos de \(z\), desativando alguns neurônios quando \(z \leq 0\), tornando os cálculos computacionais mais fáceis.
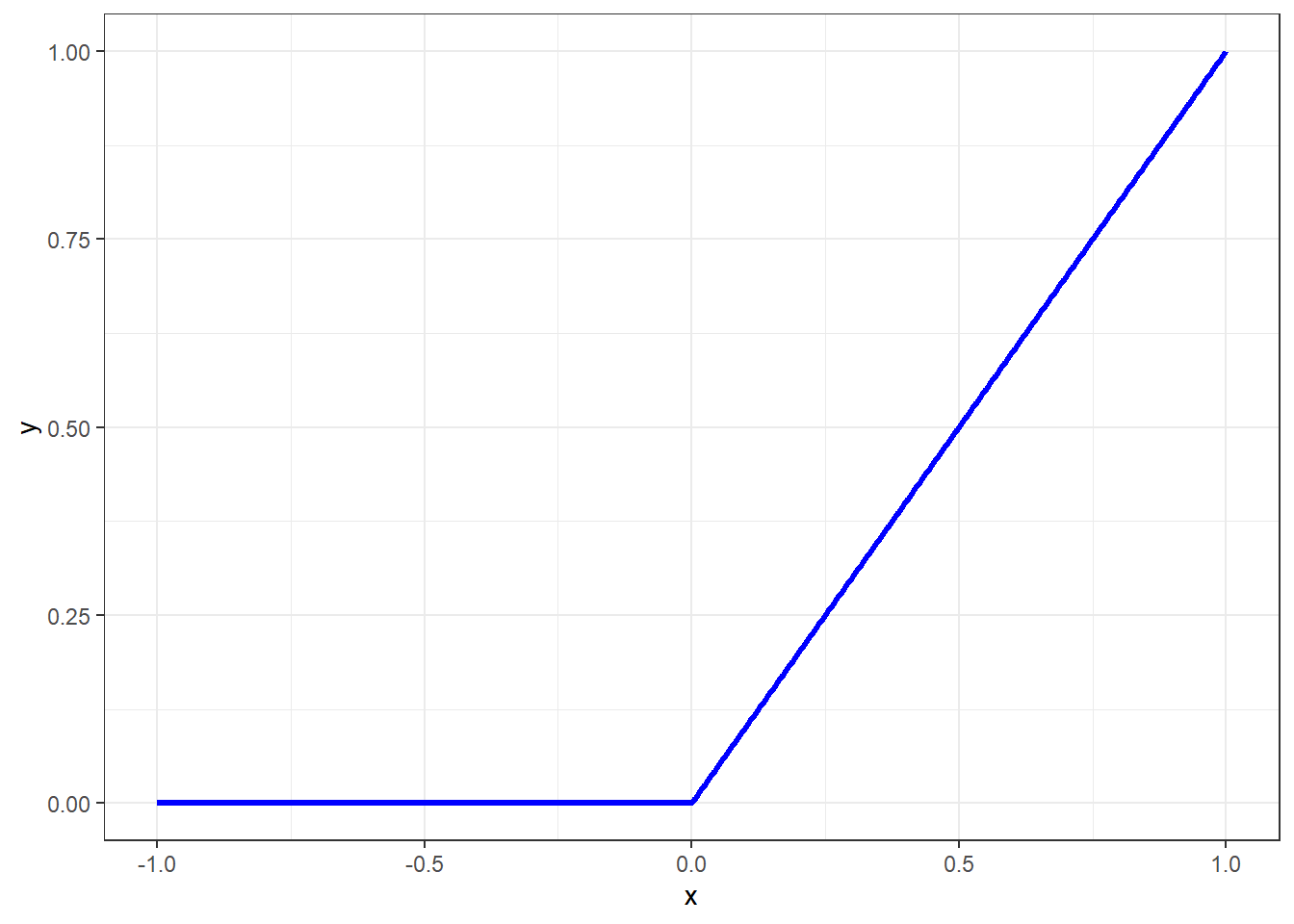
É importante esclarecer que a função ReLU deve ser utilizada em camadas ocultas, conforme a descrição matemática aqui sugere. Ademais em problemas de regressão não se usa uma função de ativação na camada de saída, ainda de acordo com a formulação aqui exposta. Oportunamente, quando o tema classificação for abordado, outras funções de ativação serão expostas, além de adaptações necessárias.
Dadas tais considerações, pode-se escrever o modelo explícito para uma rede neural com uma camada oculta conforme segue. \[ \begin{aligned} f(\mathbf{x})=v_0 + v_1.g(w_{10} + w_{11}x_1 + w_{12}x_2 + \ldots + w_{1K}x_K)\\ + v_2.g(w_{20} + w_{21}x_1 + w_{22}x_2 + \ldots + w_{2K}x_K)\\ + \ldots + v_P.g(w_{P0} + w_{P1}x_1 + w_{P2}x_2 + \ldots + w_{PK}x_K) \end{aligned} \]
Logo, \[ \begin{matrix} f(\mathbf{x})=v_0 + v_1.g(w_{10} + \sum_{k=1}^Kw_{1k}x_k) + v_2.g(w_{20} + \sum_{k=1}^Kw_{2k}x_k) + \ldots \\ + v_P.g(w_{P0} + \sum_{k=1}^Kw_{Pk}x_k) \end{matrix} \]
De forma, mais compacta, temos o modelo já exposto anteriormente. \[ \begin{matrix} f(\mathbf{x})=v_0 + \sum_{p=1}^Pv_p.g(w_{p0} + \sum_{k=1}^Kw_{pk}x_k)\\ f(\mathbf{x})=v_0 + \sum_{p=1}^Pv_ph_p(\mathbf{x}) = \mathbf{v}^T\mathbf{h}\\ \end{matrix} \]
O aprendizado profundo consiste simplesmente em uma rede neural com duas ou mais camadas ocultas.
11.2 Descida do gradiente e propagação para trás
Os parâmetros a serem estimados do modelo de rede neural consistem em \(\mathbf{v} = [v_0, v_1,v_2,\ldots,v_p]^T\) e \(\mathbf{w}_p=[w_{p0},w_{p1},w_{p2},\ldots,w_{pK}]^T\). Tomando as \(N\) observações de treino \((\mathbf{x}_i,y_i)\), \(i=1,...,N\), estima-se o modelo minimizando a função perda quadrática da Equação 11.4. \[ \min_{\{w_p\}_1^N,v} \frac{1}{2} \sum_{i=1}^{N}(y_i-f(\mathbf{x}_i))^2 \tag{11.4}\]
onde
\[ f(\mathbf{x})=v_0+\sum_{p=1}^Pv_p.g(w_{p0}+\sum_{k=1}^Kw_{pk}x_{ik}). \]
Devido a não convexidade da função perda, múltiplas soluções podem estar presentes. A otimização é realizada de forma lenta e iterativa com o algoritmo de descida do gradiente. Seja \(\mathbf{\theta}\) o vetor de todos os parâmetros da rede, \(\mathbf{\theta} = [\mathbf{v}, \mathbf{w}_0, \mathbf{w}_1, ..., \mathbf{w}_P]^T\). A fução perda pode ser reescrita conforme Equação 11.5. \[ L(\theta)=\frac{1}{2} \sum_{i=1}^{N}(y_i-f_\theta(\mathbf{x}_i))^2 \tag{11.5}\]
O algoritmo de descida do gradiente pode ser descrito a partir dos seguintes passos:
- Defina um valor inicial \(\theta_0\) para \(\theta\).
- Itere até \(L(\theta)\) parar de decrescer ou então até um critério de parada:
- Encontre um vetor \(\delta\) de forma que \(\theta_{t+1}=\theta_t+\delta\) reduza \(L(\theta)\).
- Faça \(t \leftarrow t+1\).
O valor de \(\delta\) deve ser escolhido na direção de máximo crescimento da função perda. O algoritmo de propagação para trás (backpropagation) considera o gradiente da função perda em relação aos parâmetros da rede, isto é, \[ \nabla L(\theta_t)=\frac{\partial L}{\partial \theta} \Bigg|_{\theta=\theta_t}. \]
Como \(\nabla L(\theta_t)\), que consiste no vetor de derivadas parciais de \(L\) avaliadas em \(\theta_t\), o algoritmo de descida do gradiente busca mover o vetor \(\theta\) na direção contrária do gradiente, considerando uma taxa de aprendizagem \(\rho\) pequena para facilitar a convergência, isto é, \[ \theta_{t+1} \leftarrow \theta_t - \rho \nabla L(\theta_t) \]
A propagação para trás consiste simplesmente na aplicação da regra da cadeia de diferenciação. Como \[ L(\theta)=\sum_{i=1}^NL_i(\theta)=\frac{1}{2} \sum_{i=1}^{N}(y_i-f_\theta(\mathbf{x}_i))^2 \] é uma soma, o gradiente também será uma soma em \(N\). Assim, a derivada pode ser computada termo a termo, para cada observação \(i\): \[ L_i=\frac{1}{2} (y_i-v_0-\sum_{p=1}^Pv_p.g(w_{p0}+\sum_{k=1}^Kw_{pk}x_{ik}))^2. \]
Considerando o termo \(v_p\), a derivada parcial da função perda pela regra da cadeia fica conforme segue:
\[ \frac{\partial L_i}{\partial v_p}=\frac{\partial L_i}{\partial f_\theta}\frac{\partial f_\theta}{\partial v_p} \]
\[ \frac{\partial L_i}{\partial v_p}=-(y_i-f_\theta(\mathbf{x}_i)).g(w_{p0}+\sum_{k=1}^Kw_{pk}x_{ik}). \]
Já derivando em relação ao termo \(w_{pk}\), tem-se: \[ \frac{\partial L_i}{\partial w_{pk}}=\frac{\partial L_i}{\partial f_\theta}\frac{\partial f_\theta}{\partial g(w_{p0}+\sum_{k=1}^Kw_{pk}x_{ik})}\frac{\partial g(w_{p0}+\sum_{k=1}^Kw_{pk}x_{ik})}{\partial (w_{p0}+\sum_{k=1}^Kw_{pk}x_{ik})} \frac{\partial (w_{p0}+\sum_{k=1}^Kw_{pk}x_{ik})}{\partial w_{pk}} \]
\[ \frac{\partial L_i}{\partial w_{pk}}=-(y_i-f_\theta(\mathbf{x}_i))v_pg'(w_{p0}+\sum_{k=1}^Kw_{pk}x_{ik})x_{ik}. \]
Pode-se observar que em ambos resultados das derivadas os resíduos \(y_i-f_\theta(\mathbf{x}_i)\) estão presentes. Ou seja, na diferenciação uma fração do resíduos é atribuída aos parâmetros a partir da regra da cadeia.
Dada a possibilidade de overfitting no processo de otimização dos parâmetros da rede, diversas estratégias são usadas. Por exemplo a regularização ou penalização rígida ou LASSO pode ser aplicada, sendo a função perda modificada conforme Equação 11.6 para o primeiro caso. \[ L(\theta)=\frac{1}{2} \sum_{i=1}^{N}(y_i-f_\theta(\mathbf{x}))^2 +\lambda\sum_p\theta_p^2 \tag{11.6}\]
Outra estratégia utilizada é o dropout, que consiste na remoção aleatória de uma proporção dos neurônios de uma ou mais camadas. Este processo tem, de certa forma, similaridade com a estratégia de regularização via LASSO e também com a estratégia de seleção de variáveis para o particionamento binário recursivo no algoritmo de floresta aleatória.
O processo de treinamento da rede neural envolve a definição da arquitetura da rede, isto é, o número de camadas ocultas e o número de neurônios em cada uma, além da otimização dos hiperparâmetros de encolhimento e dropout. Todos estes podem ser considerados hiperpâmetros a serem otimizados e, para tal, deve-se utilizar de validação cruzada e grid search.
Existem diversos outros tipos de redes neurais, como as redes neurais convolucionais, com grande potencial para classificação de imagens e as redes neurais recorrentes, para problemas de séries temporais, reconhecimento de fala, entre outros. O leitor é convidado a ler a bibliografia citada para mais informações.
11.3 Implementação de uma rede neural para regressão passo a passo na linguagem R
A seguir será implementada uma rede neural com algoritmo de propagação para trás, passo a passo.
set.seed(123)
# Bibliotecas necessárias
library(ggplot2)
library(gridExtra)
theme_set(theme_bw())Inicialmente define-se a função de ativação ReLU.
# Função ReLU: g(z) = max(0, z)
relu <- function(z) {
result <- pmax(0, z)
# Garantir que mantenha as dimensões originais
if (is.matrix(z)) {
result <- matrix(result, nrow = nrow(z), ncol = ncol(z))
}
return(result)
}
# Derivada da ReLU: g'(z) = 1 se z > 0, 0 caso contrário
relu_derivative <- function(z) {
result <- as.numeric(z > 0)
# Garantir que mantenha as dimensões originais
if (is.matrix(z)) {
result <- matrix(result, nrow = nrow(z), ncol = ncol(z))
}
return(result)
}Em sequência são gerados dados fictícios para regressão, com duas variáveis independentes e a resposta.
# Gerar dados
n <- 1000
x1 <- runif(n, -2, 2)
x2 <- runif(n, 0, 4)
noise <- rnorm(n, 0, 0.1)
# Função verdadeira: y = 0.3*x1 + sqrt(x2) + ruído
y_true <- 0.3 * x1 + sqrt(x2)
y <- y_true + noise
# Padronizar as variáveis para melhor convergência
x1_scaled <- scale(x1)[,1]
x2_scaled <- scale(x2)[,1]
y_scaled <- scale(y)[,1]
# Dividir em treino (80%) e teste (20%)
treino <- sample(1:n, 0.8 * n)
x1_train <- x1_scaled[treino]
x2_train <- x2_scaled[treino]
y_train <- y_scaled[treino]
x1_test <- x1_scaled[-treino]
x2_test <- x2_scaled[-treino]
y_test <- y_scaled[-treino]A rede neural e o algortmo de propagação para trás e treinamento são implementados à seguir. Inicialmente defini-se a função da rede neural. As dimensões da camada de entrada, da camada oculta e da taxa de aprendizagem devem ser dadas ao executar a função. A inicialização dos pesos é realizada via ninicialização normal de Xavier sorteando valores na distribuição normal com média nula e desvio padrão igual a \(\sqrt{2/K+1}\) e para os pesos de entrada e da camada oculta respectivamente. No treinamento, o passo à frente simplesmente avalia a rede com os pesos atuais. O passo atrás calcula o erro ou resíduo na saída, calcula o gradiente e propaga o erro para a camada oculta. Em sequência os pesos são atualizados considerando a taxa de aprendizagem e calcula-se a função perda. O procedimento é repetido até alcançar o critério de parada, sendo este o número de iterações (epochs) definido.
# Classe para Rede Neural
NeuralNetwork <- function(input_size = 2, hidden_size = 8, learning_rate = 0.01) {
# Inicializacao dos pesos (Xavier)
w <- matrix(rnorm(hidden_size * (input_size + 1), 0, sqrt(2/(input_size + 1))),
nrow = hidden_size, ncol = input_size + 1)
v <- matrix(rnorm(hidden_size + 1, 0, sqrt(2/(hidden_size + 1))), ncol = 1)
# Passo a frente
forward <- function(X) {
# Garantir que X seja matriz
if (is.vector(X)) {
X <- matrix(X, nrow = 1)
}
# X deve ser uma matriz n x 2 (x1, x2)
n_samples <- nrow(X)
# Adicionar bias (coluna de 1s) à entrada
X_bias <- cbind(1, X)
# Camada oculta: h_p = g(w_p0 + w_p1*x1 + w_p2*x2)
z_hidden <- X_bias %*% t(w) # n x hidden_size
h_hidden <- relu(z_hidden) # aplicar ReLU
# vicio na camada oculta
h_bias <- cbind(1, h_hidden)
# Camada de saida: f(x) = v0 + sum(v_p * h_p)
output <- h_bias %*% v
return(list(output = output, h_hidden = h_hidden, z_hidden = z_hidden, X_bias = X_bias))
}
# Propagacao para tras
backward <- function(X, y_true, forward_result) {
n_samples <- nrow(X)
# Resultados passo a frente
y_pred <- forward_result$output
h_hidden <- forward_result$h_hidden
z_hidden <- forward_result$z_hidden
X_bias <- forward_result$X_bias
# Erro saida
error_output <- y_pred - y_true
# Gradientes para v (pesos da camada de saida)
h_bias <- cbind(1, h_hidden)
grad_v <- t(h_bias) %*% error_output / n_samples
# Gradientes para w (pesos da camada oculta)
# Propagacao do erro para a camada oculta
error_hidden <- (error_output %*% t(v[-1])) * relu_derivative(z_hidden)
grad_w <- t(error_hidden) %*% X_bias / n_samples
return(list(grad_v = grad_v, grad_w = grad_w))
}
# Função de perda (MSE)
compute_loss <- function(X, y_true) {
forward_result <- forward(X)
y_pred <- forward_result$output
mse <- mean((y_pred - y_true)^2)
return(mse)
}
# Treinamento
train <- function(X_train, y_train, X_val = NULL, y_val = NULL, epochs = 1000, verbose = F) {
train_losses <- numeric(epochs)
val_losses <- numeric(epochs)
for (epoch in 1:epochs) {
# passo a frente
forward_result <- forward(X_train)
# passo a frente
gradients <- backward(X_train, y_train, forward_result)
# atualizar pesos
v <<- v - learning_rate * gradients$grad_v
w <<- w - learning_rate * gradients$grad_w
# calcular perdas
train_loss <- compute_loss(X_train, y_train)
train_losses[epoch] <- train_loss
if (!is.null(X_val)) {
val_loss <- compute_loss(X_val, y_val)
val_losses[epoch] <- val_loss
}
# progresso
if (verbose && epoch %% 100 == 0) {
cat(sprintf("Epoch %d: Perda treino = %.6f", epoch, train_loss))
if (!is.null(X_val)) {
cat(sprintf(", MSE validacao = %.6f", val_loss))
}
cat("\n")
}
}
return(list(train_losses = train_losses, val_losses = val_losses))
}
# Predicao
predict <- function(X) {
forward_result <- forward(X)
return(forward_result$output)
}
#
list(
forward = forward,
backward = backward,
compute_loss = compute_loss,
train = train,
predict = predict,
get_weights = function() list(w = w, v = v)
)
}A seguir é realizado o treinamento considerando s dados separados para treino. Sugere-se testar outros valores de número de neurônios na camada oculta (hidden_size), taxa de aprendizagem e epochs.
# Criar e treinar a rede neural
cat("Criando rede neural com 1 camada oculta e 8 neurônios...\n")Criando rede neural com 1 camada oculta e 8 neurônios...nn <- NeuralNetwork(input_size = 2, hidden_size = 8, learning_rate = 0.01)
# Preparar dados de treino
X_train <- cbind(x1_train, x2_train)
X_test <- cbind(x1_test, x2_test)
# Debug: verificar dimensoes
cat("Dim dados:\n")Dim dados:cat(sprintf("X_train: %d x %d\n", nrow(X_train), ncol(X_train)))X_train: 800 x 2cat(sprintf("y_train: %d\n", length(y_train)))y_train: 800cat(sprintf("X_test: %d x %d\n", nrow(X_test), ncol(X_test)))X_test: 200 x 2cat(sprintf("y_test: %d\n", length(y_test)))y_test: 200# Treinar o modelo
cat("Iniciando treino...\n")Iniciando treino...history <- nn$train(X_train, y_train, X_test, y_test, epochs = 2000, verbose = F)Em seguida avalia-se o desempenho do modelo obtido.
# Fazer predicoes
y_pred_train <- nn$predict(X_train)
y_pred_test <- nn$predict(X_test)
# Calcular metricas
rmse_train <- sqrt(mean((y_pred_train - y_train)^2))
rmse_test <- sqrt(mean((y_pred_test - y_test)^2))
r2_train <- 1 - sum((y_train - y_pred_train)^2)/sum((y_train - mean(y_train))^2)
r2_test <- 1 - sum((y_test - y_pred_test)^2)/sum((y_test - mean(y_test))^2)
cat("\n=== RESULTADOS ===\n")
=== RESULTADOS ===cat(sprintf("RMSE Treino: %.4f\n", rmse_train))RMSE Treino: 0.2013cat(sprintf("RMSE Teste: %.4f\n", rmse_test))RMSE Teste: 0.2036cat(sprintf("R2 Treino: %.4f\n", r2_train))R2 Treino: 0.9588cat(sprintf("R2 Teste: %.4f\n", r2_test))R2 Teste: 0.9606Alguns gráficos são implementados conforme segue.
# Grafico 1: Curva de aprendizado
epochs <- 1:length(history$train_losses)
loss_data <- data.frame(
Epoch = rep(epochs, 2),
Loss = c(history$train_losses, history$val_losses),
Type = rep(c("Treino", "Validação"), each = length(epochs))
)
p1 <- ggplot(loss_data, aes(x = Epoch, y = Loss, color = Type)) +
geom_line(size = 1) +
labs(y = "Perda (MSE)", color ="") +
scale_color_manual(values = c("Treino" = "blue", "Validação" = "red")) +
scale_linetype_manual(values = c("Treino" = 1, "Validação" = 3)) +
ylim(0,0.2)Warning: Using `size` aesthetic for lines was deprecated in ggplot2 3.4.0.
ℹ Please use `linewidth` instead.# Grafico 2: Predicoes vs Observacoes (Treino)
pred_data_train <- data.frame(
Real = y_train,
Predito = as.vector(y_pred_train)
)
p2 <- ggplot(pred_data_train, aes(x = Real, y = Predito)) +
geom_point(alpha = 0.2, color = "blue") +
geom_abline(intercept = 0, slope = 1, color = "red", linetype = "dashed") +
labs(subtitle = paste("Treino (R² =", round(r2_train, 3), ")"),
x = "Observado")
# Grafico 3: Predicoes vs Observacoes (Teste)
pred_data_test <- data.frame(
Real = y_test,
Predito = as.vector(y_pred_test)
)
p3 <- ggplot(pred_data_test, aes(x = Real, y = Predito)) +
geom_point(alpha = 0.2, color = "darkgreen") +
geom_abline(intercept = 0, slope = 1, color = "red", linetype = "dashed") +
labs(subtitle = paste("Teste (R2 =", round(r2_test, 3), ")"),
x = "Observado")
# Gráfico 4: Grafico de contorno
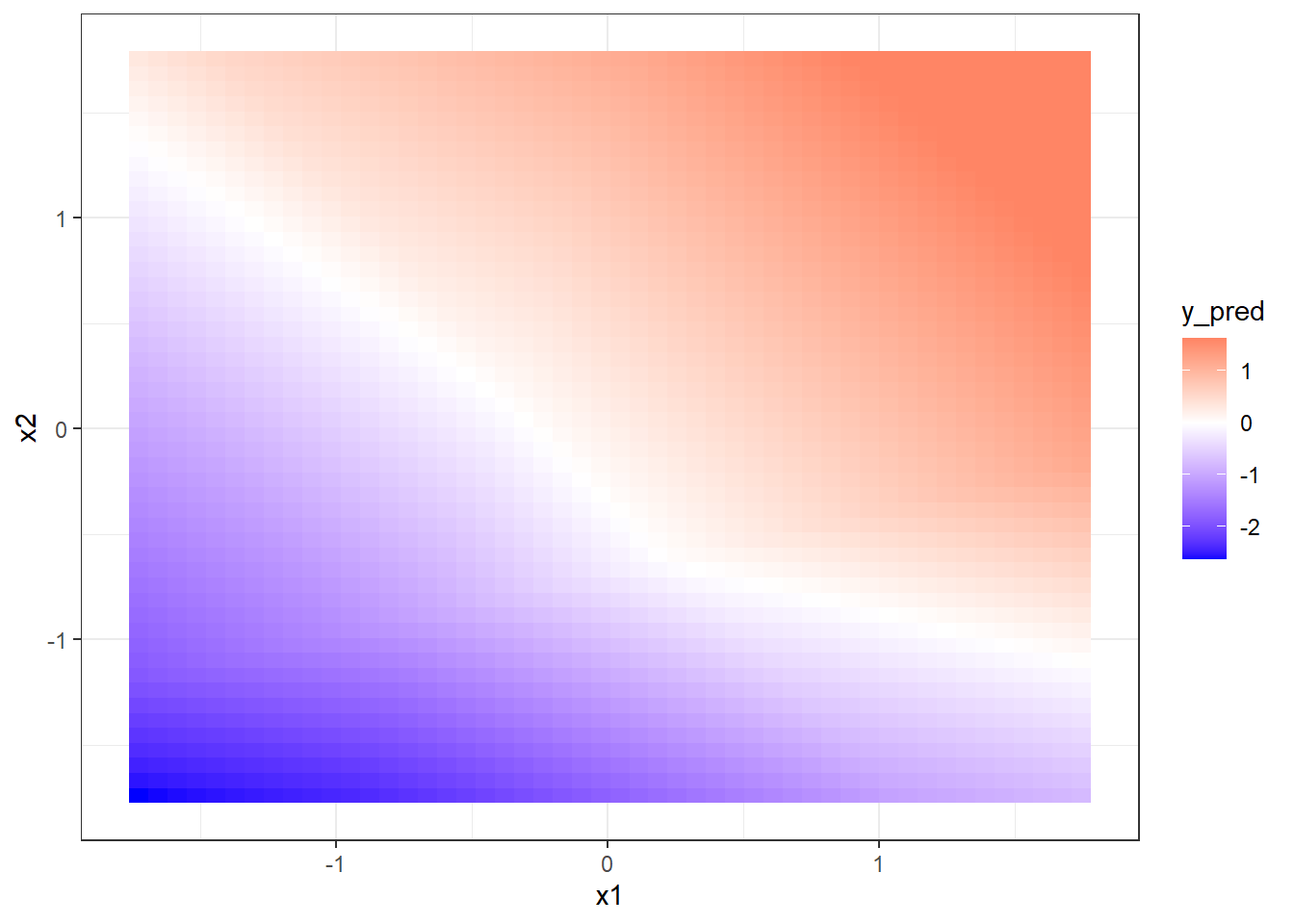
x1_grid <- seq(min(x1_scaled), max(x1_scaled), length.out = 50)
x2_grid <- seq(min(x2_scaled), max(x2_scaled), length.out = 50)
grid <- expand.grid(x1 = x1_grid, x2 = x2_grid)
X_grid <- as.matrix(grid)
y_grid <- nn$predict(X_grid)
grid$y_pred <- as.vector(y_grid)
p4 <- ggplot(grid, aes(x = x1, y = x2, fill = y_pred)) +
geom_tile() +
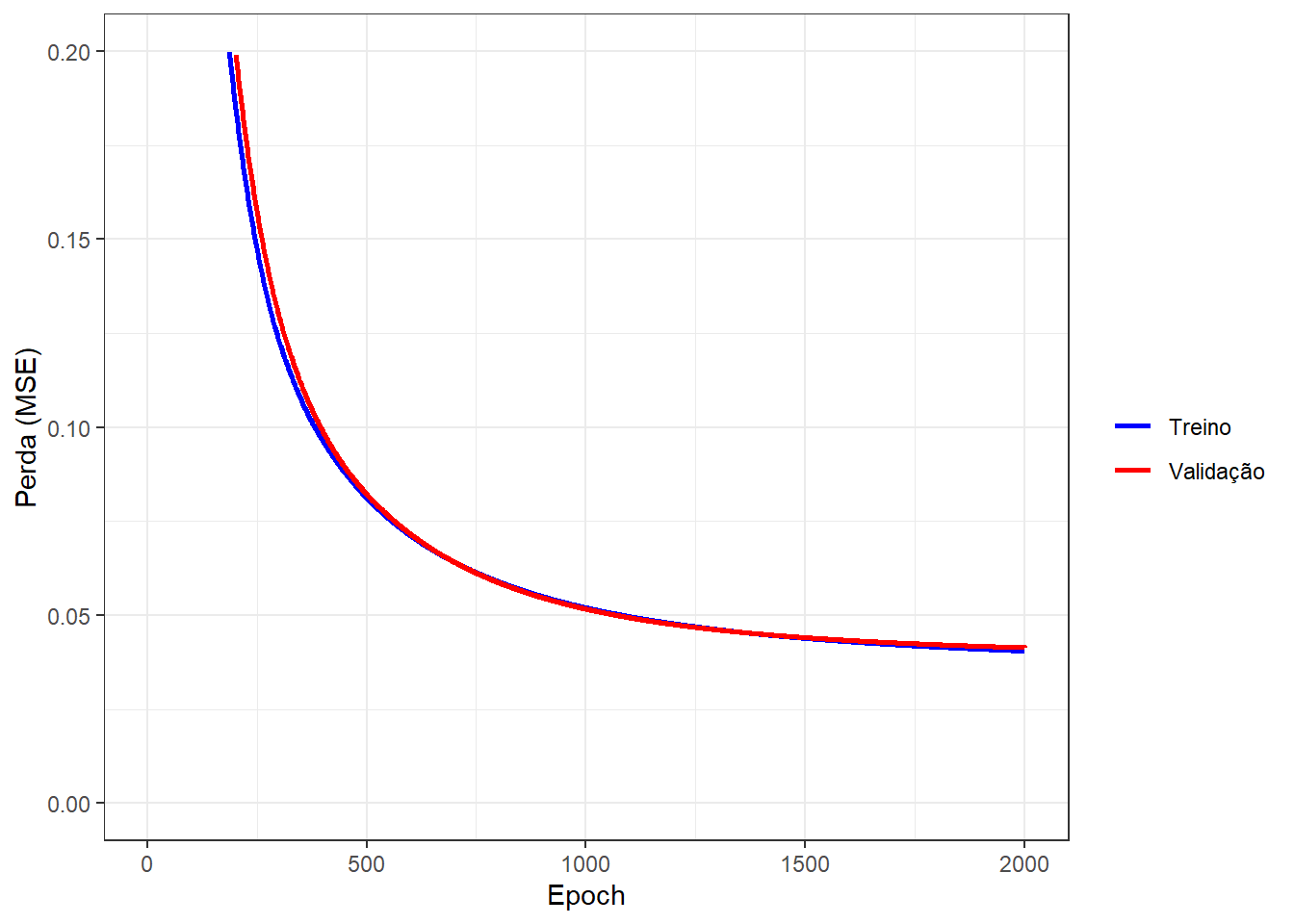
scale_fill_gradient2(low = "blue", mid = "white", high = "red", midpoint = 0)A Figura 11.2 plota a evolução da função perda para os dados de treino e de teste.
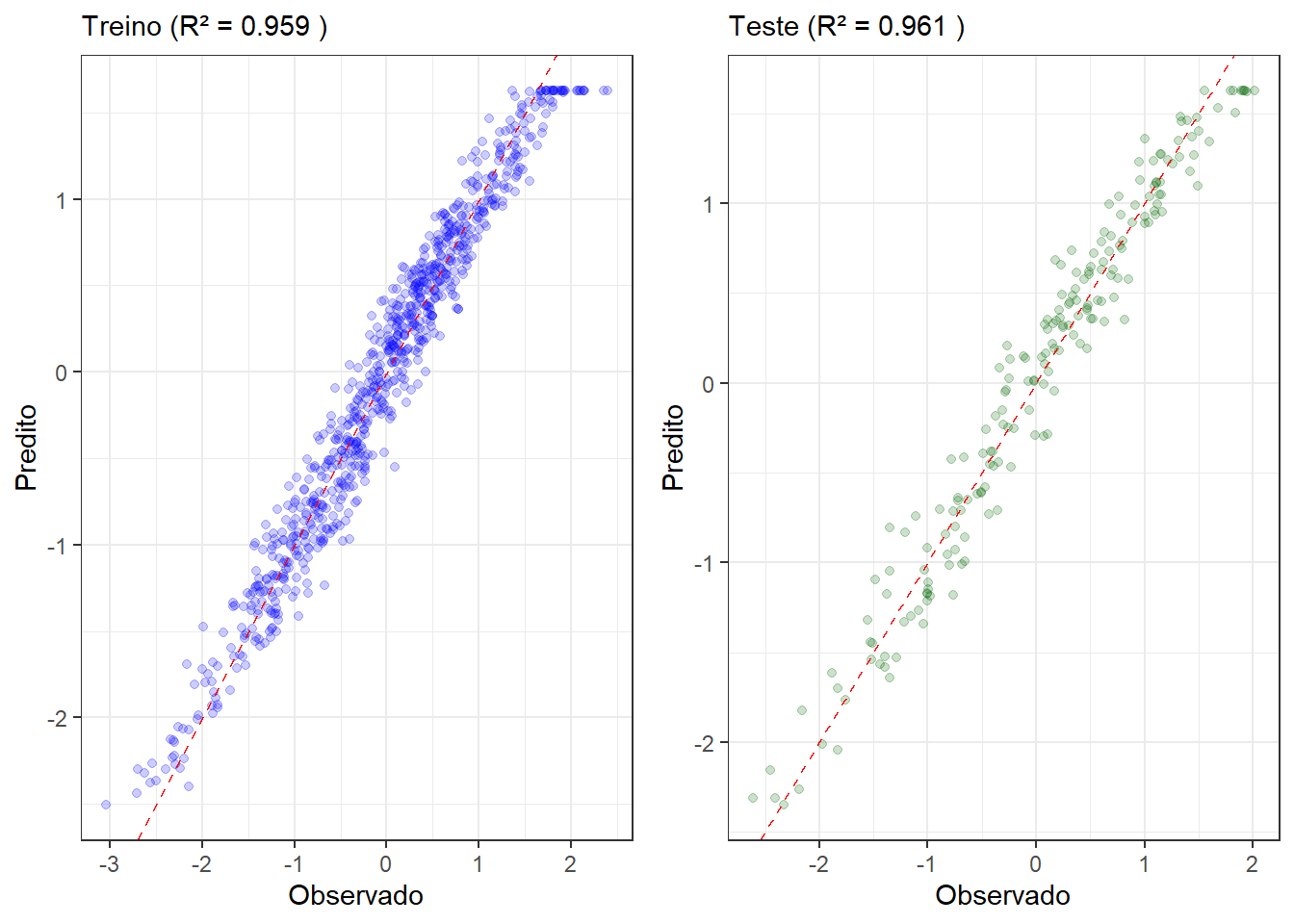
A Figura 11.3 plota as previsões versus as observações para os dados de treino e de teste.
A Figura 11.4 plota o gráfico de contorno para a rede neural obtida.
Aqui é realizada uma comparação com um modelo de regressão linear múltipla.
# Ajustar modelo linear para comparação
lm_model <- lm(y_train ~ x1_train + x2_train)
y_pred_lm_train <- predict(lm_model)
y_pred_lm_test <- predict(lm_model, newdata = data.frame(x1_train = x1_test, x2_train = x2_test))
rmse_lm_train <- sqrt(mean((y_pred_lm_train - y_train)^2))
rmse_lm_test <- sqrt(mean((y_pred_lm_test - y_test)^2))
r2_lm_train <- summary(lm_model)$r.squared
r2_lm_test <- 1 - sum((y_test - y_pred_lm_test)^2)/sum((y_test - mean(y_test))^2)
cat("Rede Neural:\n")Rede Neural:cat(sprintf(" RMSE Teste: %.4f, R2 Teste: %.4f\n", rmse_test, r2_test)) RMSE Teste: 0.2036, R2 Teste: 0.9606cat("Regressão Linear:\n")Regressão Linear:cat(sprintf(" RMSE Teste: %.4f, R2 Teste: %.4f\n", rmse_lm_test, r2_lm_test)) RMSE Teste: 0.2487, R2 Teste: 0.9412Por fim, são apresentados os pesos ótimos obtidos para a rede.
weights <- nn$get_weights()
cat("\n=== PESOS DA REDE NEURAL ===\n")
=== PESOS DA REDE NEURAL ===cat("Pesos da camada oculta (w):\n")Pesos da camada oculta (w):print(round(weights$w, 4)) x1_train x2_train
[1,] 1.9242 -0.8116 -0.6826
[2,] -0.5910 -0.3866 -0.2250
[3,] -0.1675 -0.8945 -0.6355
[4,] -0.3704 0.0516 -0.0623
[5,] -1.2383 -0.2510 -0.6065
[6,] -2.1539 -0.1231 -0.5666
[7,] -0.5086 -1.2340 0.3557
[8,] -0.8132 0.3516 -1.1909cat("\nPesos da camada de saída (v):\n")
Pesos da camada de saída (v):print(round(weights$v, 4)) [,1]
[1,] 1.6279
[2,] -0.7421
[3,] 0.3169
[4,] -0.3611
[5,] 0.1283
[6,] 0.3160
[7,] 0.5798
[8,] 0.1635
[9,] -0.613511.4 Validação cruzada e grid search para treinar múltiplos modelos de regressão passo a passo via tidymodels
Previsão do preço de computadores.
library(Ecdat) # para dados
library(tidymodels)
library(modelsummary)
library(finetune) # para grid search
library(dplyr)
library(baguette) # bag_treeLeitura de dados.
data(Computers)
dados2 <- na.omit(Computers)Entendendo os dados.
dados2 |> glimpse()Rows: 6,259
Columns: 10
$ price <dbl> 1499, 1795, 1595, 1849, 3295, 3695, 1720, 1995, 2225, 2575, 21…
$ speed <dbl> 25, 33, 25, 25, 33, 66, 25, 50, 50, 50, 33, 66, 50, 25, 50, 50…
$ hd <dbl> 80, 85, 170, 170, 340, 340, 170, 85, 210, 210, 170, 210, 130, …
$ ram <dbl> 4, 2, 4, 8, 16, 16, 4, 2, 8, 4, 8, 8, 4, 8, 8, 4, 2, 4, 4, 8, …
$ screen <dbl> 14, 14, 15, 14, 14, 14, 14, 14, 14, 15, 15, 14, 14, 14, 14, 14…
$ cd <fct> no, no, no, no, no, no, yes, no, no, no, no, no, no, no, no, n…
$ multi <fct> no, no, no, no, no, no, no, no, no, no, no, no, no, no, no, no…
$ premium <fct> yes, yes, yes, no, yes, yes, yes, yes, yes, yes, yes, yes, yes…
$ ads <dbl> 94, 94, 94, 94, 94, 94, 94, 94, 94, 94, 94, 94, 94, 94, 94, 94…
$ trend <dbl> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1,…Estatísticas descritivas.
datasummary_skim(dados2)| Unique | Missing Pct. | Mean | SD | Min | Median | Max | Histogram | |
|---|---|---|---|---|---|---|---|---|
| price | 808 | 0 | 2219.6 | 580.8 | 949.0 | 2144.0 | 5399.0 |  |
| speed | 6 | 0 | 52.0 | 21.2 | 25.0 | 50.0 | 100.0 |  |
| hd | 59 | 0 | 416.6 | 258.5 | 80.0 | 340.0 | 2100.0 |  |
| ram | 6 | 0 | 8.3 | 5.6 | 2.0 | 8.0 | 32.0 |  |
| screen | 3 | 0 | 14.6 | 0.9 | 14.0 | 14.0 | 17.0 |  |
| ads | 34 | 0 | 221.3 | 74.8 | 39.0 | 246.0 | 339.0 |  |
| trend | 35 | 0 | 15.9 | 7.9 | 1.0 | 16.0 | 35.0 |  |
| N | % | |||||||
| cd | no | 3351 | 53.5 | |||||
| yes | 2908 | 46.5 | ||||||
| multi | no | 5386 | 86.1 | |||||
| yes | 873 | 13.9 | ||||||
| premium | no | 612 | 9.8 | |||||
| yes | 5647 | 90.2 |
Criando coluna com combinações das variáveis categóricas que tem níveis desbalanceados.
dados2 <- dados2 |>
mutate(multi_premium = paste(multi, premium,
sep = '_'))Separando dados de treino e teste.
set.seed(16)
dados_split2 <- initial_split(dados2,
prop = 0.25,
strata = multi_premium)
dados_train2 <- training(dados_split2)
dados_test2 <- testing(dados_split2)
set.seed(17)
dados_folds2 <-
vfold_cv(v = 10, dados_train2, repeats = 2)Definindo a receita. A receita cntém o modelo, além dos preprocessamentos realizados nas variáveis preditoras.
normalized_rec2 <-
recipe(price ~ speed + hd + ram + screen + cd + multi + premium + ads + trend,
data = dados_train2) |>
step_normalize(all_numeric_predictors()) |>
step_dummy(all_nominal_predictors()) Definindo os métodos de regressão a serem testados. Deve-se definir os hiperparâmetros a serem testados e o pacote (engine), uma vez que geralmente há várias opções de pacotes em R para um mesmo método.
linear_reg_spec <-
linear_reg(penalty = tune(), mixture = tune()) |>
set_engine("glmnet")
tree_spec <- decision_tree(tree_depth = tune(), min_n = tune(), cost_complexity = tune()) |>
set_engine("rpart") |>
set_mode("regression")
bag_cart_spec <-
bag_tree(tree_depth = tune(), min_n = tune(), cost_complexity = tune()) |>
set_engine("rpart") |>
set_mode("regression")
rforest_spec <- rand_forest(mtry = tune(), min_n = tune(), trees = tune()) |>
set_engine("ranger") |>
set_mode("regression")
xgb_spec <- # evolution of GBM
boost_tree(tree_depth = tune(), learn_rate = tune(), loss_reduction = tune(),
min_n = tune(), sample_size = tune(), trees = tune()) |>
set_engine("xgboost") |>
set_mode("regression")
svm_r_spec <-
svm_rbf(cost = tune(), rbf_sigma = tune()) |>
set_engine("kernlab") |>
set_mode("regression")
svm_p_spec <-
svm_poly(cost = tune(), degree = tune()) |>
set_engine("kernlab") |>
set_mode("regression")
mars_spec <- # method similar to GAM
mars(prod_degree = tune()) %>%
set_engine("earth") %>%
set_mode("regression")
nnet_spec <-
mlp(hidden_units = tune(), penalty = tune(), epochs = tune()) |>
set_engine("nnet", MaxNWts = 2600) |>
set_mode("regression")
nnet_param <-
nnet_spec |>
extract_parameter_set_dials() |>
update(hidden_units = hidden_units(c(1, 27)))Definindo o workflow. O workflow contém a receita e os modelos.
normalized2 <-
workflow_set(
preproc = list(normalized = normalized_rec2),
models = list(linear_reg = linear_reg_spec,
tree = tree_spec,
bagging = bag_cart_spec,
rforest = rforest_spec,
XGB = xgb_spec,
SVM_radial = svm_r_spec,
SVM_poly = svm_p_spec,
MARS = mars_spec,
neural_network = nnet_spec)
)
normalized2# A workflow set/tibble: 9 × 4
wflow_id info option result
<chr> <list> <list> <list>
1 normalized_linear_reg <tibble [1 × 4]> <opts[0]> <list [0]>
2 normalized_tree <tibble [1 × 4]> <opts[0]> <list [0]>
3 normalized_bagging <tibble [1 × 4]> <opts[0]> <list [0]>
4 normalized_rforest <tibble [1 × 4]> <opts[0]> <list [0]>
5 normalized_XGB <tibble [1 × 4]> <opts[0]> <list [0]>
6 normalized_SVM_radial <tibble [1 × 4]> <opts[0]> <list [0]>
7 normalized_SVM_poly <tibble [1 × 4]> <opts[0]> <list [0]>
8 normalized_MARS <tibble [1 × 4]> <opts[0]> <list [0]>
9 normalized_neural_network <tibble [1 × 4]> <opts[0]> <list [0]>all_workflows2 <-
bind_rows(normalized2) |>
# Make the workflow ID's a little more simple:
mutate(wflow_id = gsub("(simple_)|(normalized_)", "", wflow_id))
all_workflows2# A workflow set/tibble: 9 × 4
wflow_id info option result
<chr> <list> <list> <list>
1 linear_reg <tibble [1 × 4]> <opts[0]> <list [0]>
2 tree <tibble [1 × 4]> <opts[0]> <list [0]>
3 bagging <tibble [1 × 4]> <opts[0]> <list [0]>
4 rforest <tibble [1 × 4]> <opts[0]> <list [0]>
5 XGB <tibble [1 × 4]> <opts[0]> <list [0]>
6 SVM_radial <tibble [1 × 4]> <opts[0]> <list [0]>
7 SVM_poly <tibble [1 × 4]> <opts[0]> <list [0]>
8 MARS <tibble [1 × 4]> <opts[0]> <list [0]>
9 neural_network <tibble [1 × 4]> <opts[0]> <list [0]>Realizando grid search e validação cruzada.
race_ctrl <-
control_race(
save_pred = TRUE,
parallel_over = "everything",
save_workflow = TRUE
)
race_results2 <-
all_workflows2 |>
workflow_map(
"tune_race_anova",
seed = 1503,
resamples = dados_folds2,
grid = 25,
control = race_ctrl
)→ A | warning: A correlation computation is required, but `estimate` is constant and has 0
standard deviation, resulting in a divide by 0 error. `NA` will be returned.There were issues with some computations A: x1There were issues with some computations A: x6There were issues with some computations A: x9There were issues with some computations A: x16There were issues with some computations A: x23There were issues with some computations A: x26There were issues with some computations A: x27
There were issues with some computations A: x27Extraindo métricas para avaliar os resultados da validação cruzada.
collect_metrics(race_results2) |>
filter(.metric == "rmse") |>
arrange(mean)# A tibble: 37 × 9
wflow_id .config preproc model .metric .estimator mean n std_err
<chr> <chr> <chr> <chr> <chr> <chr> <dbl> <int> <dbl>
1 XGB Preproce… recipe boos… rmse standard 173. 20 3.20
2 rforest Preproce… recipe rand… rmse standard 197. 20 3.80
3 SVM_poly Preproce… recipe svm_… rmse standard 213. 20 3.60
4 bagging Preproce… recipe bag_… rmse standard 214. 20 4.10
5 bagging Preproce… recipe bag_… rmse standard 216. 20 4.63
6 SVM_radial Preproce… recipe svm_… rmse standard 219. 20 4.58
7 MARS Preproce… recipe mars rmse standard 230. 20 3.47
8 tree Preproce… recipe deci… rmse standard 247. 20 5.51
9 neural_network Preproce… recipe mlp rmse standard 259. 20 11.6
10 neural_network Preproce… recipe mlp rmse standard 261. 20 7.00
# ℹ 27 more rowscollect_metrics(race_results2) |>
filter(.metric == "rsq") |>
arrange(desc(mean))# A tibble: 37 × 9
wflow_id .config preproc model .metric .estimator mean n std_err
<chr> <chr> <chr> <chr> <chr> <chr> <dbl> <int> <dbl>
1 XGB Preproce… recipe boos… rsq standard 0.912 20 0.00315
2 rforest Preproce… recipe rand… rsq standard 0.887 20 0.00331
3 SVM_poly Preproce… recipe svm_… rsq standard 0.866 20 0.00511
4 bagging Preproce… recipe bag_… rsq standard 0.865 20 0.00420
5 bagging Preproce… recipe bag_… rsq standard 0.862 20 0.00444
6 SVM_radial Preproce… recipe svm_… rsq standard 0.859 20 0.00515
7 MARS Preproce… recipe mars rsq standard 0.844 20 0.00454
8 tree Preproce… recipe deci… rsq standard 0.819 20 0.00802
9 neural_network Preproce… recipe mlp rsq standard 0.798 20 0.0108
10 neural_network Preproce… recipe mlp rsq standard 0.795 20 0.0206
# ℹ 27 more rowsVisualizando desempenho dos métodos.
IC_rmse2 <- collect_metrics(race_results2) |>
filter(.metric == "rmse") |>
group_by(wflow_id) |>
filter(mean == min(mean)) |>
group_by(wflow_id) |>
arrange(mean) |>
ungroup()
IC_r22 <- collect_metrics(race_results2) |>
filter(.metric == "rsq") |>
group_by(wflow_id) |>
filter(mean == max(mean)) |>
group_by(wflow_id) |>
arrange(desc(mean)) |>
ungroup()
IC2 <- bind_rows(IC_rmse2, IC_r22)
ggplot(IC2, aes(x = factor(wflow_id, levels = unique(wflow_id)), y = mean)) +
facet_wrap(~.metric, scales = "free") +
geom_point(stat="identity", aes(color = wflow_id), pch = 1) +
geom_errorbar(stat="identity", aes(color = wflow_id,
ymin=mean-1.96*std_err,
ymax=mean+1.96*std_err), width=.2) +
labs(y = "", x = "method") + theme_bw() +
theme(legend.position = "none",
axis.text.x = element_text(angle = 90, vjust = 0.5, hjust=1))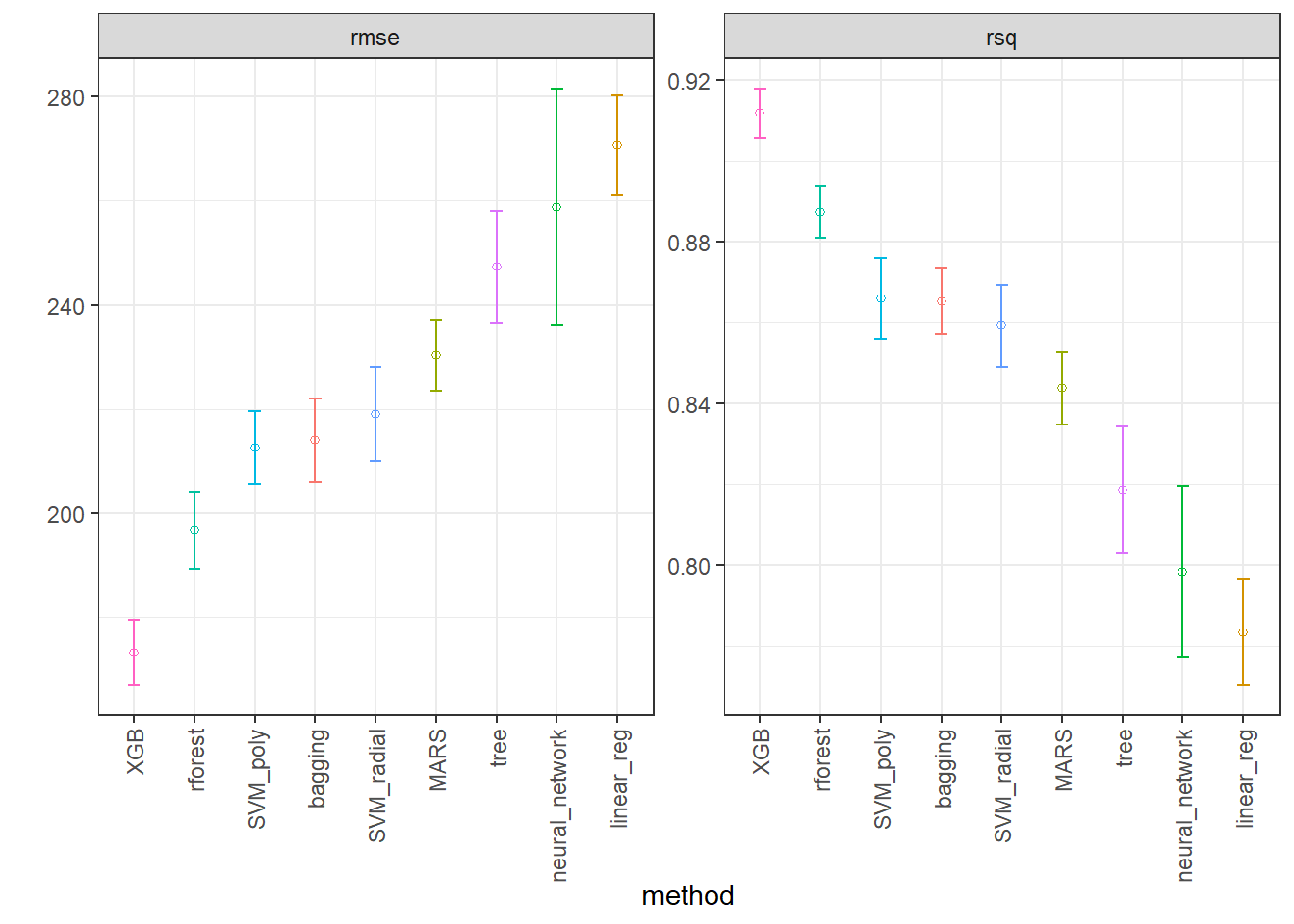
Obtendo níveis ótimos dos hiperparâmetros do melhor modelo.
best_rmse2 <-
race_results2 |>
extract_workflow_set_result("XGB") |>
select_best(metric = "rmse")
best_rmse2# A tibble: 1 × 7
trees min_n tree_depth learn_rate loss_reduction sample_size .config
<int> <int> <int> <dbl> <dbl> <dbl> <chr>
1 1416 9 5 0.0226 3.48 0.962 Preprocessor1_Mo…XGB_test_results <-
race_results2 |>
extract_workflow("XGB") |>
finalize_workflow(best_rmse2) |>
last_fit(split = dados_split2)
collect_metrics(XGB_test_results)# A tibble: 2 × 4
.metric .estimator .estimate .config
<chr> <chr> <dbl> <chr>
1 rmse standard 170. Preprocessor1_Model1
2 rsq standard 0.915 Preprocessor1_Model1Plotando previstos versus observados para os dados de teste dos dois melhores métodos.
best_rmse2_2 <-
race_results2 |>
extract_workflow_set_result("rforest") |>
select_best(metric = "rmse")
best_rmse2_2# A tibble: 1 × 4
mtry trees min_n .config
<int> <int> <int> <chr>
1 6 1167 2 Preprocessor1_Model17rf_test_results <-
race_results2 |>
extract_workflow("rforest") |>
finalize_workflow(best_rmse2_2) |>
last_fit(split = dados_split2)
collect_metrics(rf_test_results)# A tibble: 2 × 4
.metric .estimator .estimate .config
<chr> <chr> <dbl> <chr>
1 rmse standard 196. Preprocessor1_Model1
2 rsq standard 0.888 Preprocessor1_Model1test_results2 <- rbind(XGB_test_results |>
collect_predictions(),
rf_test_results |>
collect_predictions())
test_results2$method <- c(rep("XGB", nrow(XGB_test_results |>
collect_predictions())),
rep("rforest", nrow(rf_test_results |>
collect_predictions())))
test_results2 |>
ggplot(aes(x = price, y = .pred)) +
geom_abline(color = "gray50", lty = 2) +
geom_point(alpha = 0.2) +
facet_grid(col = vars(method)) +
coord_obs_pred() +
labs(x = "observed", y = "predicted") +
theme_bw()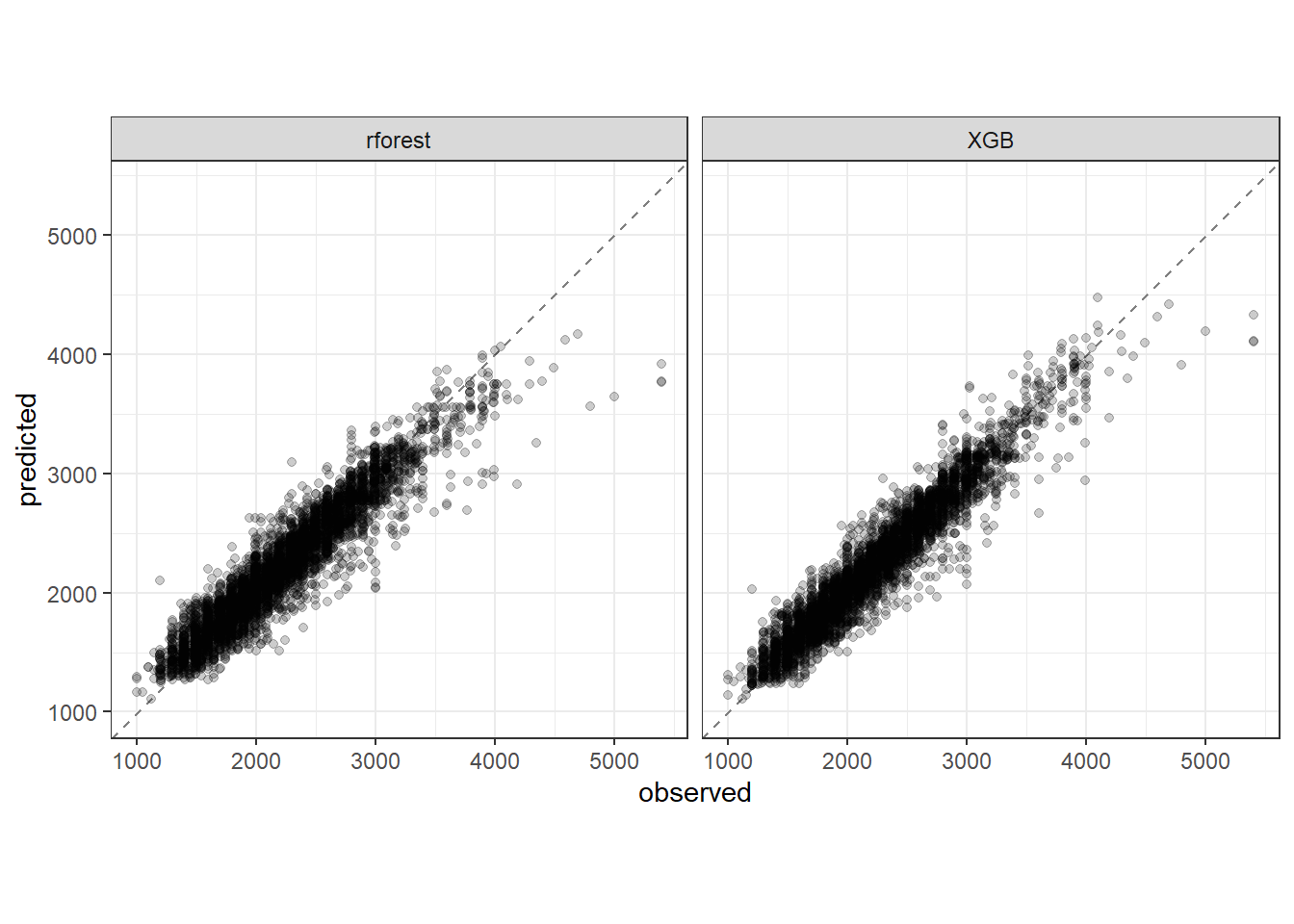
Modelo final.
XGB_final <- race_results2 |>
extract_workflow("XGB") |>
finalize_workflow(best_rmse2)
XGB_final══ Workflow ════════════════════════════════════════════════════════════════════
Preprocessor: Recipe
Model: boost_tree()
── Preprocessor ────────────────────────────────────────────────────────────────
2 Recipe Steps
• step_normalize()
• step_dummy()
── Model ───────────────────────────────────────────────────────────────────────
Boosted Tree Model Specification (regression)
Main Arguments:
trees = 1416
min_n = 9
tree_depth = 5
learn_rate = 0.0226030302714192
loss_reduction = 3.48070058842842
sample_size = 0.9625
Computational engine: xgboost Referências
Bishop, Christopher M., and Hugh Bishop. “Deep learning: foundations and concepts.” (2024).
Gareth, J., Daniela, W., Trevor, H., & Robert, T. (2013). An introduction to statistical learning: with applications in R. Spinger.